LIS-C
Responsable : Romain Nattier
Présentation
Sur le plan théorique, le LIS cherche à faire une synthèse des approches descriptives et évolutives de la systématique. Il s’agit de définir ce qui est commun et différent dans les concepts, les structures, les méthodes, utilisés par la taxonomie, la phylogénétique et la biogéographie.Il a ainsi été montré l’unicité et la cohérence d’application de la méthode cladistique à trois éléments pour la phylogénie et la biogéographie comparative.
Dans sa composante de modélisation des connaissances descriptives en systématique (observations morpho-anatomiques et hypothèses d’homologie/caractères) la réflexion théorique rejoint le développement des ontologies en informatique (modèle) et en biologie (contenus).
La théorie propose des modèles et des contraintes qui doivent pouvoir être confrontés à des cas d’application pour en évaluer la validité et les limites. Pour cela le LIS investit largement, en aval de la recherche théorique, dans le développement de logiciels. Chacun de ces logiciels cible une ou des tâches nécessaires à la taxonomie, la phylogénétique et la biogéographie : Xper, dans ses différentes versions mono-utilisateur ou collaborative, est dédié à l’informatisation et à l’analyse des descriptions taxonomiques (étape descriptive de la systématique) ; Lisbeth implémente l’analyse cladistique à trois éléments utilisée en phylogénie et en biogéographie comparative (cladistique).
Quelques travaux :

Une plateforme collaborative et d’expertise sur l’identification taxonomique (Xper3).
Combiner l’humain et l’automatique dans la reconnaissance des espèces
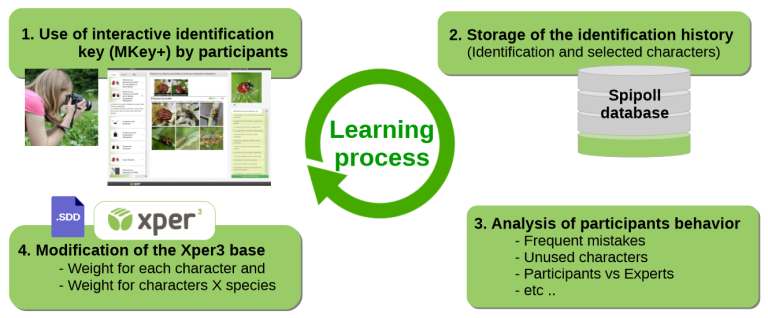
Notre projet Xper-ease vise à concevoir comment combiner de façon intelligente des compétences humaines (observation de traits, ou réalisation de mesures) et des méthodes automatiques, dans un outil d’aide à l’identification. L’analyse d’images peut aussi s’avérer particulièrement utile pour des caractères continus difficiles à exprimer par des états discrets.
Un travail préliminaire sur l’utilisation des réseaux de neurones pour extraire des traits morphologiques dans des images d’herbier a été réalisé en 2017 en collaboration avec le LIP6, la Direction des collections du MNHN et l’IRD.

Poster présenté au congrès TDWG en 2017.
Histoire des noms et base de données
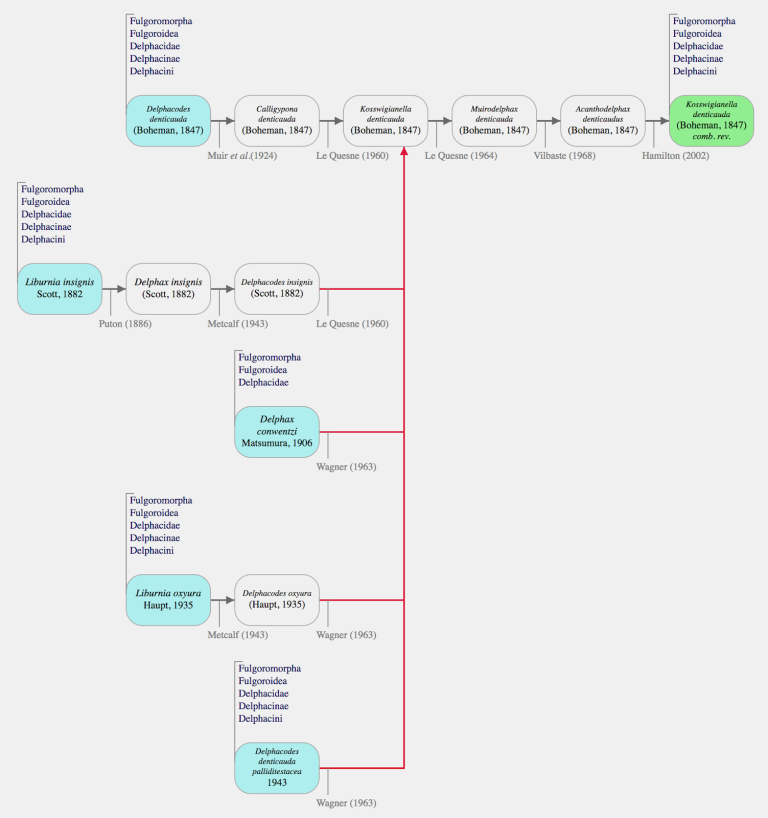
Ecran extrait de la base de données FLOW. Visualisation de l’histoire des noms.
Le projet TRASSE
La mise en œuvre est opérationnelle dans le cadre du programme de sciences participatives SPIPOLL (http://spipoll.snv.jussieu.fr/mkey/mkey-spipoll.html). Ce travail contribuera aux objectifs du projet d’envergure « 65 Millions d’observateurs » financé par le programme d’investissement d’avenir pour 2015-2018 (https://65mo.wordpress.com/ ).
Vers la liste du personnel LIS
Vers la rubrique des thèses en cours
VERS LA RUBRIQUE DES THESES SOUTENUES






